DNA-viivakoodaus mahdollisti uusia tutkimustapoja, jotka DNA-metaviivakoodaus vie vielä astetta pidemmälle.
Molekulaariset menetelmät ovat mullistaneet ekologisen tutkimuksen viimeisen viidenkymmenen vuoden aikana. Eräs näistä menetelmistä on molekulaarinen lajintunnistus, jonka eri sovellutuksilla voidaan parhaimmillaan tunnistaa biologista materiaalia lajilleen DNA:n perusteella. Yksittäiseen, kullekin lajiryhmälle tähän tarkoitukseen valittuun DNA-alueeseen perustuvaa lajintunnistusta kutsutaan DNA-viivakoodaukseksi, joka terminä on vakiintunut jo oppimateriaaleihinkin.
DNA-viivakoodaus toimii (valtaosassa eliöryhmistä) varsin mallikkaasti, ja sen käytännön sovelluksiin on herätty viimeisen parinkymmenen vuoden aikana niin tutkimuksessa kuin teollisuudessa. Nykyään esimerkiksi Ruokaviraston epäilemän kasvitautiesiintymän, sushissa käytetyn kalalajin tai perhosharrastajan tuntemattomaksi jääneen näyteyksilön tunnistaminen molekulaarisesti on mahdollista, jopa helppoa. Tutkimuspuolella tuntomerkkien avulla tunnistetun näytteen lajinmäärityksen varmistaminen DNA-menetelmillä on jo arkipäivää. Varsinkin jos kyse on suurista näytemääristä, on DNA-viivakoodaus myös verrattain halpaa.
Perinteisen DNA-viivakoodauksen rajoitteet
Perinteisellä DNA-viivakoodauksella on kuitenkin teoreettiset ja tekniset rajoitteensa. Valittu DNA-viivakoodialue on aina kompromissi, eikä sen erotuskyky ole kaikissa ryhmissä tai lajipareissa riittävä tarkkaan lajintunnistukseen. Joissain ryhmissä voidaan joutua tyytymään suku- tai heimotason tunnistukseen. Lisäksi jos vertailtavaa sekvenssiaineistoa ei ole, ei näytteen yhdistäminen lajinimeen onnistu. Viivakoodialue on myös hyvin herkkä valituille alukkeille, eikä tiettyihin lajiryhmiin soveltuvia alukkeita ole läheskään aina tarjolla.
DNA-viivakoodaus perustuu DNA:n eristämisen jälkeiseen PCR:ään, jota seuraa klassinen sangersekvensointi. Tunnistettavan näytteen DNA:n pitää siis olla puhdasta, ehjää ja sitä tulee olla riittävästi. DNA-viivakoodaus usein epäonnistuu, jos näyte on liian vanha, säilytetty epäoptimaalisella tavalla tai kontaminoitunut vieraalla biologisella materiaalilla.
Sekanäytteet tuottavat harmaita hiuksia DNA-viivakoodauksen käyttäjille. Koska sangersekvensoinnissa luetaan vain yksi sekvensointituote, ei tällä menetelmällä voida kunnolla käsitellä useampaa eri kohdesekvenssiä sisältävää näytettä. Jos halutaan vaikka tunnistaa kasviplanktonnäytteestä kaikki planktonlajit, moniyrttivalmisteesta kaikki siinä käytetyt kasvilajit tai ihmisen suolistosta otetun näytteen kaikki bakteerilajit, ei perinteistä DNA-viivakoodausta voida käyttää. Sen sijaan viimeisen kymmenen vuoden aikana ratkaisun tähän ongelmaan on tuonut DNA-metaviivakoodaus.
Rinnakkainen metaviivakoodaus
Metaviivakoodauksessa etuliite ”meta-” viittaa rinnakkaisuuteen. Menetelmässä käytännössä suoritetaan viivakoodausta sekanäytteen kaikista lajeista rinnakkain, hyödyntäen Next Generation Sequencing (NGS) -menetelmiä sangersekvensoinnin sijaan. Metaviivakoodauksessa DNA eristetään ja monistetaan PCR:llä samalla tavalla kuin tavallisessa viivakoodauksessa. Kaikki PCR-tuotteet sekvensoidaan yhdessä NGS-sekvensointireaktiossa, ja eri PCR-tuotteet voidaan erotetella bioinformatiikan avulla sekvensoinnin jälkeen. Näin voidaan tunnistaa potentiaalisesti sekanäytteen kaikki ne lajit, joista saadaan PCR-tuote samoilla alukkeilla. Metaviivakoodauksen kohteena voi olla vaikka kaikki sienet, kaikki aitotumalliset tai kaikki nisäkkäät.
Jos perinteisen viivakoodauksen sovelluskohteet olivat moninaiset, metaviivakoodaus vie ne vielä astetta pidemmälle. Nyt voidaan molekulaarisesti tunnistaa kokonaisia eliöyhteisöjä, kuten vaikka siitepölynäytteitä, maaperän sienilajistoa tai biofilmiyhteisöjä. Omassa väitöstutkimuksessani käytän menetelmää lajien välisten vuorovaikutusten tutkimuksessa.
Parempia tuloksia huonommista näytteistä
NGS-sekvensoinnin tuottaman suuren datamäärän vuoksi metaviivakoodausta voi tehdä myös sellaisista näytteistä, joissa DNA on pilkkoutunut esimerkiksi näytteen iän vuoksi. Perinteinen DNA-viivakoodaus ei tähän pysty. Metaviivakoodaamalla voidaan siis katsoa myös ajassa taaksepäin, ja sekvensoida esimerkiksi järvien pohjasedimenttien DNAta, ja siten tutkia kasvillisuutta vuosituhansien takaa siitepölytutkimuksen rinnalla (ns. sedimentary ancient DNA eli sedaDNA).
Myös eliöiden ruokavalion tutkimus on ottanut uuden harppauksen, sillä metaviivakoodaamalla voidaan tunnistaa esimerkiksi petojen saaliseläimiä uloste- tai ruuansulatuskanavanäytteistä. Turun yliopiston tutkijat kollegoineen esimerkiksi selvittivät taannoin sudenkorentojen ruokavaliota lajitasolla menetelmää käyttäen.
Suomen ympäristökeskuksessa selvitetään parhaillaan, miten metaviivakoodausmenetelmiä voitaisiin käyttää Suomen luonnon tilan seurannassa pitkien aikasarjojen, esimerkiksi kasviplanktonseurannan tukena. Jopa ilmanäytteistä voidaan havaita paikallista lajistoa – keväällä julkaistiin tutkimus, jossa tanskalaiset tutkijat tutkivat Kööpenhaminan eläintarhan ilmasta kerättyä DNA:ta ja metaviivakoodasivat esiin niin eläintarhan eläimiä kuin alueen luonnonvaraisiakin eläimiä. Maailmanlaajuinen, Suomesta johdettu LIFEPLAN-hanke pyrkii automaattisen näytteenoton ja metaviivakoodausmenetelmien avulla havaitsemaan maailmanlaajuisesti globaalia sieni- ja hyönteislajistoa ilmasta ja maaperästä.
Tikat epifyyttien levittäjinä
Olen tarkastellut omassa tutkimuksessani lintujen ja epifyyttisten sienten, jäkälien ja sammalten välistä vuorovaikutusta. Erityisesti lahopuulla ruokailevien lintujen, kuten tikkojen, luulisi varsin luontevasti kuljettavan lahopuulla kasvavien sienten, jäkälien ja sammalten itiöitä ja leviämiskappaleita höyhenpuvussaan lahopuulta toiselle. Tutkimusnäyttöä tälle leviämisbiologiselle vuorovaikutukselle on kuitenkin hyvin vähän. Eläimiä on tutkittu paljon putkilokasvien siementen levittäjinä, mutta vähemmän tutkittujen eliöryhmien (sienet, jäkälät, sammalet) leviämisbiologia tunnetaan puutteellisesti.
Kehitimme museonäytteiksi päätyneiden tikkojen tutkimukseen menetelmän, jossa eristämme biologista materiaalia tikkojen pyrstösulista, rintahöyhenistä ja jaloista. Ensimmäisessa artikkelissa tunnistimme leviämisyksiköitä mikroskopian avulla, ja tällä hetkellä suoritetaan metaviivakoodaukseen perustuvaa lajintunnistusta.
Mikroskopian avulla voitiin tunnistaa suuri diversiteetti sienten, jäkälien ja sammalten rakenteita. Sieni-itiöt, jäkälien murumaiset leviämisyksiköt sekä sammalkappaleet olivat yleisimmin löytyneitä rakenteita. Suurimmat määrät leviämisyksiköitä löydettiin pyrstösulista. Lisäksi löytyi harvinaisempia rakenteita, kuten vapaana eläviä leviä, sukkulamatoja, rataseläimiä, piileviä sekä siitepölyhiukkasia. Mikroskopiatyöskentelyn perusteella tikkojen mukana liikkuu suuri monimuotoisuus erilaisten eliöiden leviämisrakenteita. Nähtävästi leviämisrakenteet ovat myös elinkelpoisia.
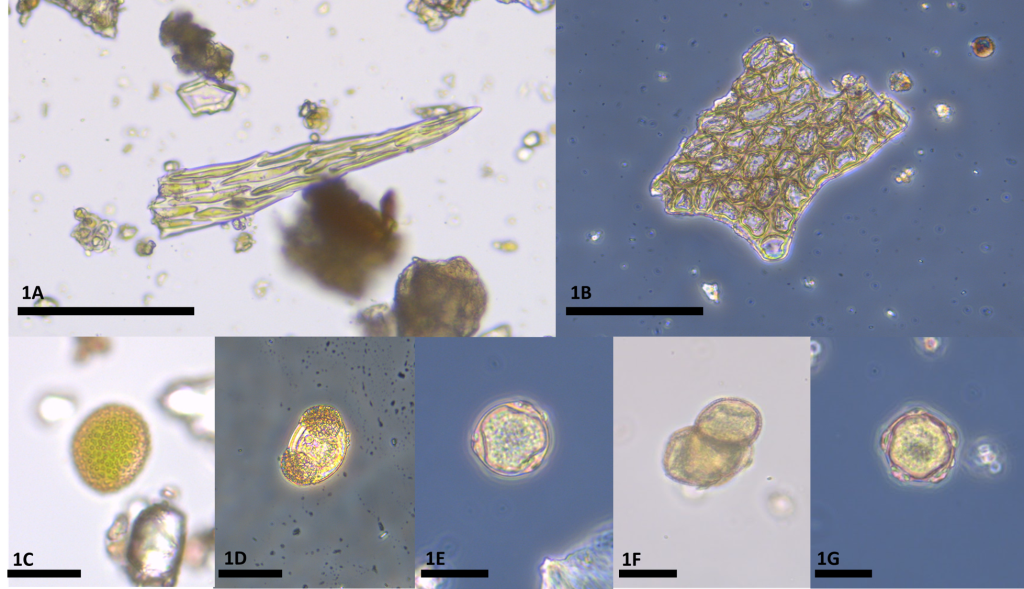
Lintujen kuljettamat sieni-itiöt
Sienten ja jäkälien lajintunnistus pelkkien itiötuntomerkkien perusteella on hankalaa. Siispä sovellamme seuraavassa tutkimuksessamme DNA-metaviivakoodausta sienten lajintunnistuksessa. Olen syysmuuton 2022 aikana laajentanut näytteenottoa eläviin lintuihin Hangon lintuasemalla. Rengastusverkkoon jäävästä linnusta otetaan itiöpyyhkäisynäytteet ennen rengastusta, joista eristetään DNA:ta laboratoriossa. Kohdelajeina ovat paitsi tikat, myös tiaiset, hippiäiset, punarinnat ja hernekertut.
PCR-vaiheiden ja sekvensoinnin jälkeen voidaan parhaimmillaan tuottaa lajilista niistä sienistä, joita lintujen mukana kulkee. Tämä avaa ovet mielenkiintoisiin ekologisiin ja evoluutiobiologisiin tutkimuskysymyksiin. Ovatko tietyt sieniryhmät yliedustettuina linnuissa? Vaikuttaako sienen kasvutapa, lisääntymisrakenne, itiökoko tai kasvupaikka asiaan? Kuljettavatko muuttolinnut materiaalia Suomeen saapuessaan tai toisaalta paikallista materiaalia pois lähtiessään?

Loputtomat sovelluskohteet
Metaviivakoodauksen ja laajemmin molekulaarisen lajintunnistuksen sovelluskohteet ekologiassa ovat loppumattomat, ja menetelmät ovat vasta vakiintumassa. Tulevaisuus tuo tullessaan varmasti uusia harppauksia menetelmissä ja uusia, yhä innovatiivisempia käyttökohteita niille. Uutta dataa tullaan tuottamaan valtavasti, teratavuittain.
Seuraava iso kysymys lienee se, miten näin suuresta määrästä dataa voidaan tehdä järkeviä ja yleistettäviä ekologisia johtopäätöksiä. Myös vertailuaineisto muodostaa ongelman. Jos tunnistuksen kohteena oleva laji on lähtökohtaisesti vieras (esim. tieteelle tuntematon kuvaamaton laji, tai laji joka tunnetaan, mutta josta ei ole vertailukelpoista sekvenssiä), ei viivakoodausmenetelmälläkään saada lajille nimeä – sekvenssi jää mysteeriksi. Museonäytteiden viivakoodaus tulevaisuudessa saattaa ratkaista ongelmaa ainakin osittain, jos sillä voidaan täydentää referenssitietokantoja kattamaan enemmän lajeja.
Usein viivakoodausmenetelmistä keskusteltaessa nousee keskusteluun ajatus, että ”perinteinen” tuntomerkkeihin liittyvä lajintuntemustaito on tulevaisuudessa täysin tarpeeton. Hoitaahan viivakoodaus kaiken lajintunnistuksen, ja biologi voi vain tuijottaa sekvenssiä koskaan edes näkemättä tutkimaansa kohdelajia. Itse olen eri mieltä. DNA-sekvenssit ovat vain yksi tapa tulkita eliölajeja – morfologian, elintapojen, evoluutiohistorian, käyttäytymisen ja monen muun ohella. Suomen kielen termi lajintuntemustaito on oiva termi siksi, että se eroaa puhtaasta mekaanisesta lajintunnistuksesta, perustui tunnistus sitten DNA:han, ulkonäkötuntomerkkeihin tai molempiin.
Todellinen lajien tuntemus on paljon enemmän kuin pelkkää lajintunnistusta. Hyvän lajintuntemustaidon omaavalla biologilla on silmää eri tuntomerkkien hoksaamiselle, tiedon yhdistämiselle eri lähteistä sekä tietotaitoa lajintunnistusmateriaalien käyttöön – eikä välttämättä pelkästään hyvä nimimuisti.
Teksti ja kuvat: Niko Johansson. Artikkeli on julkaistu Naturassa 4/2022.
Avauskuva: Luonnontieteellisen keskusmuseon käpytikkanäytteitä. Näistä linnuista etsittiin sieni-, jäkälä- ja sammalkappaleita tarttuneina pyrstöön, höyhenistöön sekä jalkoihin.
Lähteet
Johansson, N. R., Kaasalainen, U., Rikkinen, J. (2021). Woodpeckers can act as dispersal vectors for fungi plants and microorganisms. Ecology & Evolution 11(12): 7154–7163.
Joly, S., Davies, T. J., Archambault, A., Bruneau, A., Derry, A., Kember, S. W., Peres-Neto, P. Vamosi, J. & Wheeler, T. A. (2014): Ecology in the age of DNA barcoding: the resource, the promise and the challenges ahead. Molecular Ecology Resources 14(2): 221–232.
Kaunisto, K. M., Roslin, T., Sääksjärvi, I. & Vesterinen, E. (2017): Pellets of proof: First glimpse of the dietary composition of adult odonates as revealed by metabarcoding of feces. Ecology & Evolution 7(20): 8588–8598.
LIFEPLAN: a planetary inventory for life. https://www.helsinki.fi/en/projects/lifeplan.
Lynggaard, C., Bertelsen, M.F., Jensen, C. V., Johnson, M. S., Froslev, T. G., Olsen, M. T. & Bohmann, K. (2022): Airborne environmental DNA for terrestrial vertebrate community monitoring. Current Biology 32(3): 701+.
Suomen ympäristökeskus (2021): Roadmap for Implementing Environmental DNA (eDNA) and Other Molecular Monitoring Methods in Finland – Vision and Action Plan for 2022–2025.
Taberlet, P., Bonin, A., Zinger, L. & Coissac, E. (2018): Environmental DNA – For biodiversity research and monitoring. Oxford University Press, 246 s.

